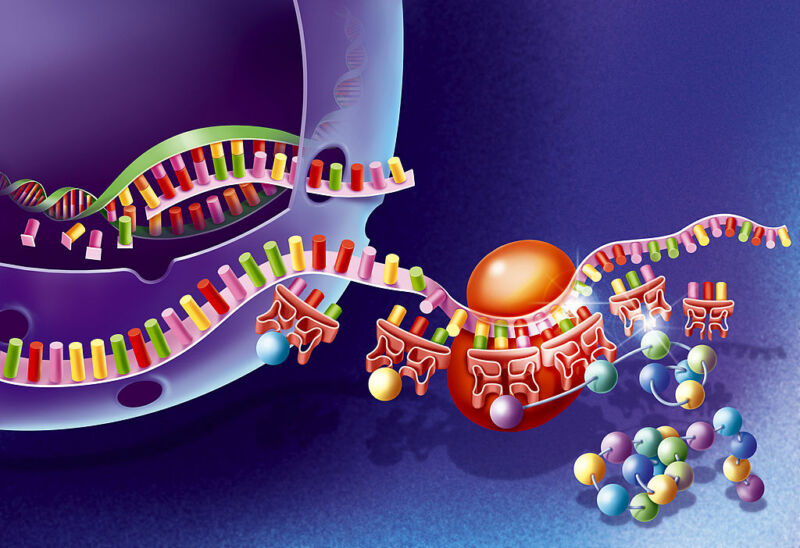
Tous les êtres vivants sur Terre utilisent une copie du même code génétique. Chaque cellule fabrique des protéines en utilisant les mêmes 20 acides aminés. Les ribosomes, la machinerie de fabrication de protéines à l’intérieur des cellules, lisent le code génétique de la molécule d’ARN messager pour décider quel acide aminé mettre ensuite dans la protéine particulière qu’ils construisent.
Ce code est universel, c’est pourquoi les ribosomes de nos cellules peuvent lire un morceau d’ARN messager viral et en faire une protéine virale fonctionnelle. Cependant, il existe de nombreux autres acides aminés. Alors que la vie ne l’utilise généralement pas, les scientifiques l’ont incorporé dans des protéines. Maintenant, les chercheurs ont trouvé un moyen d’étendre considérablement le code génétique, permettant à ces acides aminés non biologiques d’être incorporés à grande échelle. Ils y sont parvenus en activant un deuxième ensemble de tout – les protéines et les ARN – nécessaires pour traduire le code génétique.
système séparé
Les acides aminés non canoniques peuvent remplir un certain nombre de fonctions. Ils peuvent servir d’étiquettes afin que la protéine spécifique d’un chercheur puisse facilement être tracée à l’intérieur des cellules. Ils peuvent aider à réguler la fonction des protéines, permettant aux chercheurs de les activer et de les désactiver à un moment et à un endroit spécifiques de leur choix, puis de surveiller les effets éventuels. Si suffisamment de ces acides aminés non canoniques sont liés ensemble, les protéines résultantes formeront une toute nouvelle classe de biopolymères qui peuvent remplir des fonctions que les protéines conventionnelles ne peuvent pas – à des fins de recherche, thérapeutiques ou autres.
Placer des acides aminés non canoniques dans des protéines nécessite une manipulation du code génétique, pour laquelle il n’y a aucun moyen de déterminer leur utilisation. Une option est Modifier le code génétique d’une cellule, laissant la plus grande partie intacte. La variante utilise des copies modifiées de tous les composants du code génétique : ARNm orthologues, ribosomes orthologues, enzymes orthologues responsables de la lecture des ARNm et de la construction de protéines dans les ribosomes. Orthogonal signifie ici que ce mécanisme fonctionnera conjointement avec le mécanisme normal de fabrication de protéines ribosomiques dans la cellule, mais sans interférer avec celui-ci. Il ne lira et ne traduira que ses propres ARNm orthologues, pas les lignées cellulaires normales.
Ces composantes orthogonales seront impaires, elles ne sont donc pas nécessaires au fonctionnement de la cellule. Ils peuvent donc être conçus, organisés différemment et modifiés de toutes les manières que les scientifiques peuvent imaginer. Ils peuvent être utilisés pour fabriquer de nouveaux polymères et faire la lumière sur les mécanismes impliqués dans la production de protéines cellulaires normales. C’est quelque chose que nous ne pouvons pas faire avec des composants cellulaires normaux, car cela tuerait la cellule.
améliorer l’orthogonalité
Jason Chen, directeur du Centre de biologie chimique et synthétique (CCSB) à Cambridge, au Royaume-Uni, et il a fabriqué tous ces composants orthogonaux. Mais ce n’est pas très efficace. Dans un article publié cette semaine dans chimie de la nature, décrit comment je l’ai corrigé : en utilisant des algorithmes de calcul pour concevoir et optimiser les meilleurs ARNm orthologues par des ribosomes orthologues. Non seulement le rendement en protéines s’est considérablement amélioré, mais les changements ont assuré le fonctionnement efficace des ribosomes orthologues même en présence de ribosomes normaux.
« Notre compréhension des facteurs qui déterminent le rendement protéique pour la traduction normale est incomplète… Seule la moitié de la variance du rendement protéique observée peut s’expliquer par des paramètres connus », déplore l’introduction de l’ouvrage. Cependant, son laboratoire a appris que l’étape d’initiation, lorsque le ribosome attrape l’ARN messager, est une étape essentielle. Ils savaient également que la structure de l’ARNm est importante. Ils ont donc commencé à muter l’ARNm orthogonal pour changer ces deux côtés et ont sélectionné les mutations qui se lient bien aux ribosomes orthogonaux mais aux ribosomes normaux de qualité inférieure. Après des centaines de cycles de mutations, ils ont optimisé trois ARNm orthologues différents, codant pour trois protéines différentes. L’un d’eux contenait quatre acides aminés non canoniques.
Ensuite, le laboratoire a appliqué la même méthode pour améliorer les enzymes orthologues, et ils ont produit une augmentation de 33 fois du rendement en protéines ; Le système orthogonal fabrique maintenant la même quantité de protéines que le système cellulaire normal. Les cellules utilisées dans ce travail étaient bactéries coliMais le Dr Chen a utilisé un système orthogonal pour fabriquer des protéines non canoniques dans la levure, les cellules de mammifères, les vers et les mouches des fruits.
« Nous prévoyons que les opportunités découlant des méthodes d’incorporation de nombreux acides aminés non canoniques distincts augmenteront à mesure que le nombre d’acides aminés non canoniques pouvant être incorporés », écrivent lui et ses collègues. Ces algorithmes qu’ils ont développés pour concevoir des ARNm orthologues traduits efficacement devraient certainement les aider à atteindre cet objectif.
chimie de la nature 2021. EST CE QUE JE: 10.1038/s41557-021-00764-5

« Drogué des réseaux sociaux. Explorateur d’une humilité exaspérante. Nerd du café. Amical résolveur de problèmes. Évangéliste culinaire. Étudiant. »
